AI-forskningslaboratoriet DeepMind har skapat den mest omfattande kartan över humana proteiner hittills med hjälp av artificiell intelligens. Företaget, ett dotterbolag till Google-parent Alphabet, släpper data gratis, med vissa forskare som jämför den potentiella effekten av arbetet med Human Genome Project, ett internationellt försök att kartlägga varje mänsklig gen.
Proteiner är långa, komplexa molekyler som utför många uppgifter i kroppen, från att bygga vävnad till att bekämpa sjukdomar. Deras syfte dikteras av deras struktur, som viks som origami till komplexa och oregelbundna former. Att förstå hur ett protein viks hjälper till att förklara dess funktion, vilket i sin tur hjälper forskare med en rad uppgifter – från att driva grundläggande forskning om hur kroppen fungerar, till att designa nya läkemedel och behandlingar.
“Kulminationen av DeepMinds hela 10-årslivslängd”
Tidigare bestämdes strukturen för ett protein beroende av dyra och tidskrävande experiment. Men förra året visade DeepMind att det kan producera korrekta förutsägelser av ett proteins struktur med AI-programvara som heter AlphaFold. Nu släpper företaget hundratusentals förutsägelser från programmet till allmänheten.
“Jag ser detta som kulmen på DeepMinds hela 10-åriga livstid”, säger företagets VD och grundare Demis Hassabis till The Verge. ”Från början är detta vad vi tänkt göra: att göra genombrott inom AI, testa att på spel som Go och Atari, [och] tillämpa det på verkliga problem, för att se om vi kan påskynda vetenskapliga genombrott och använda de som gynnar mänskligheten. ”
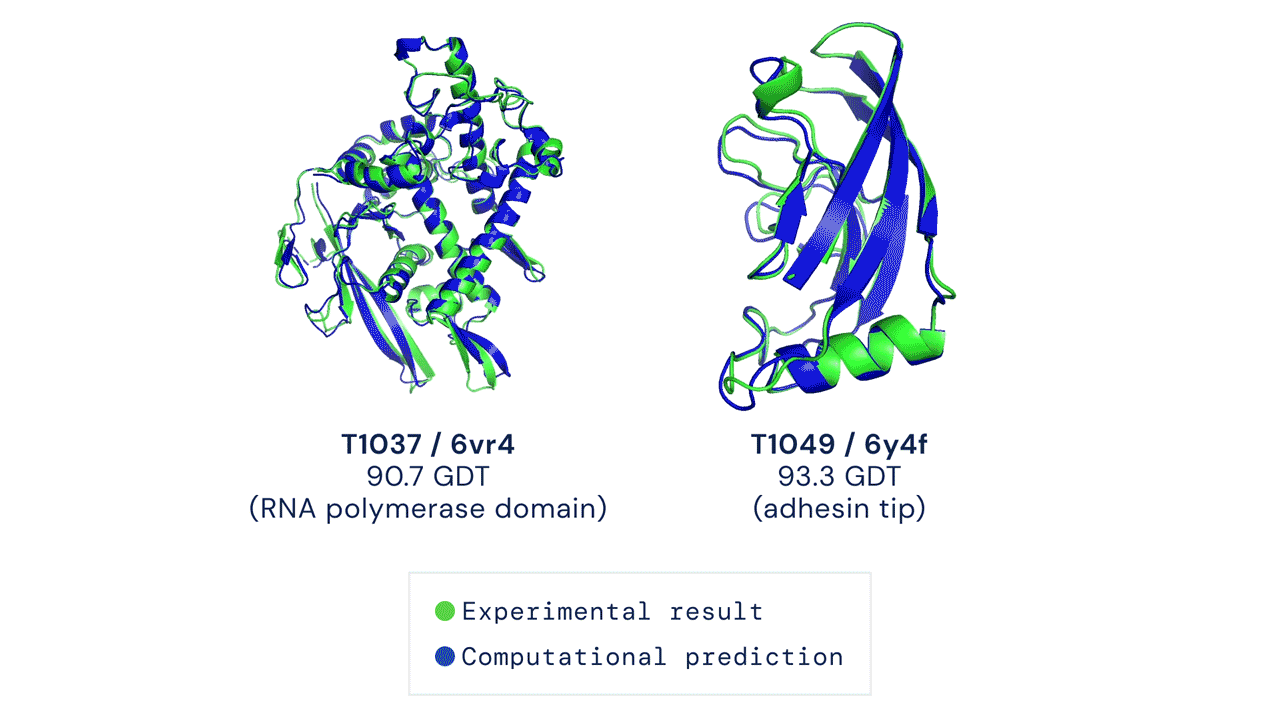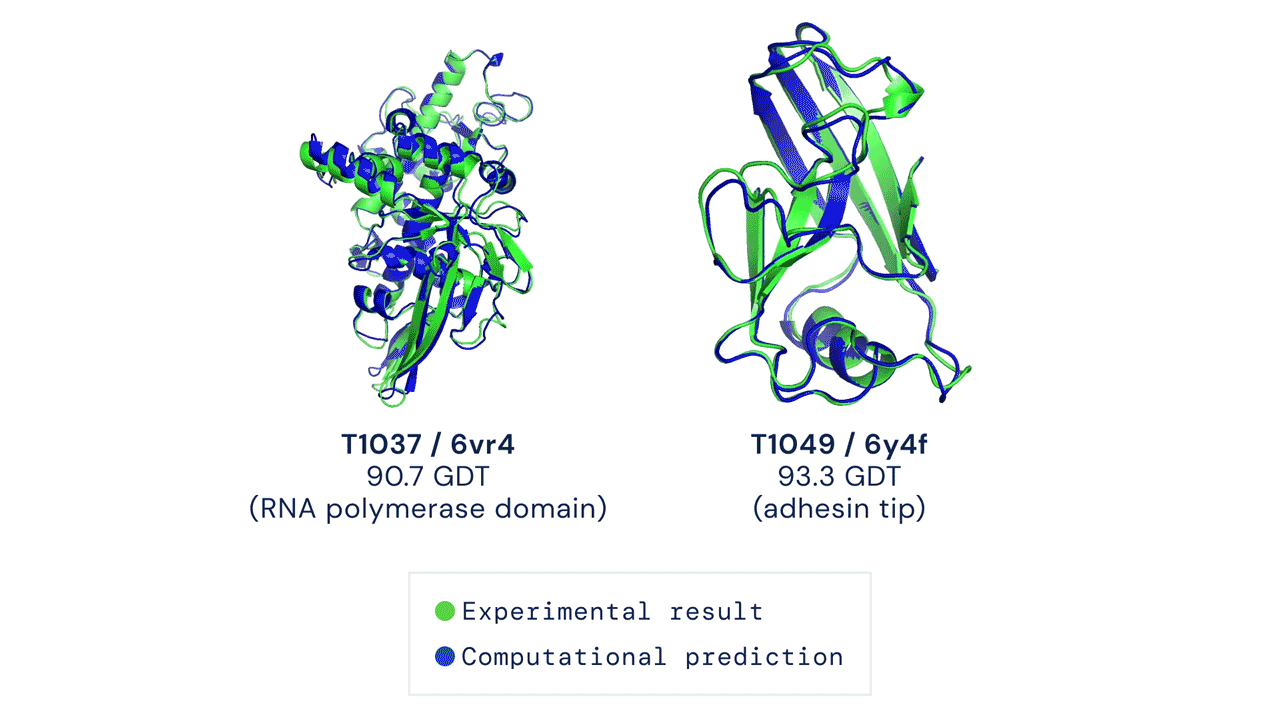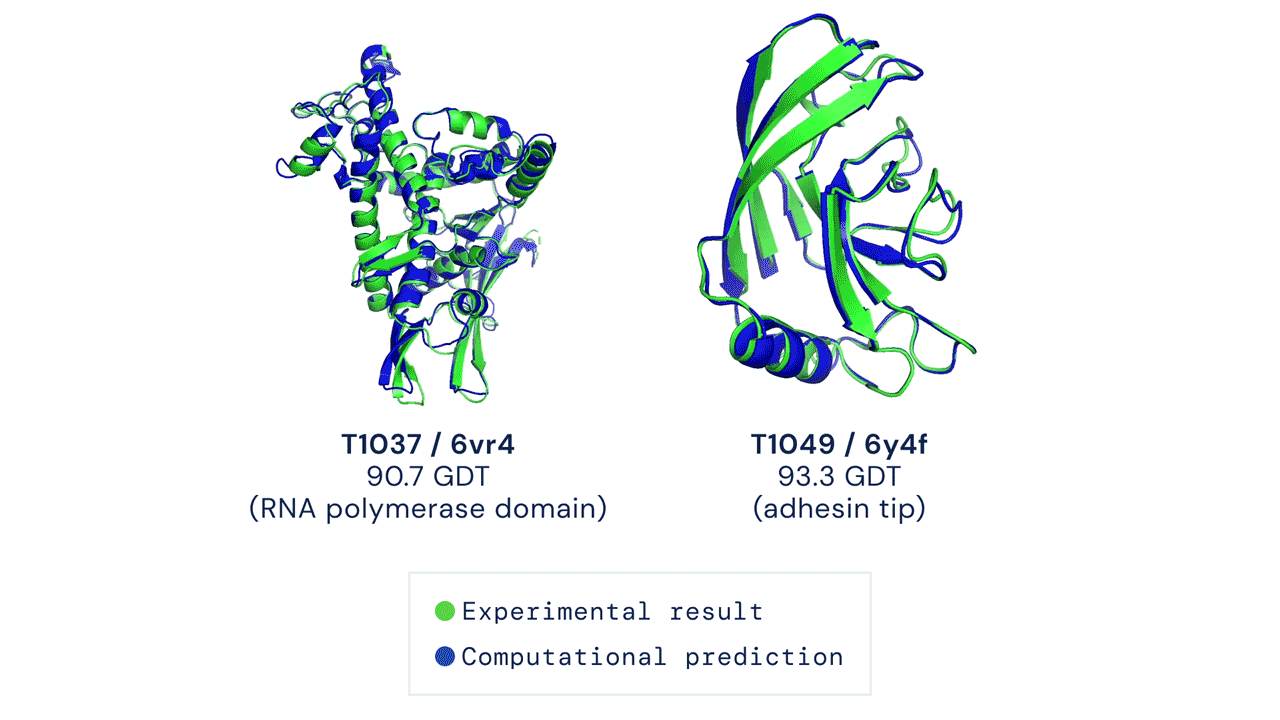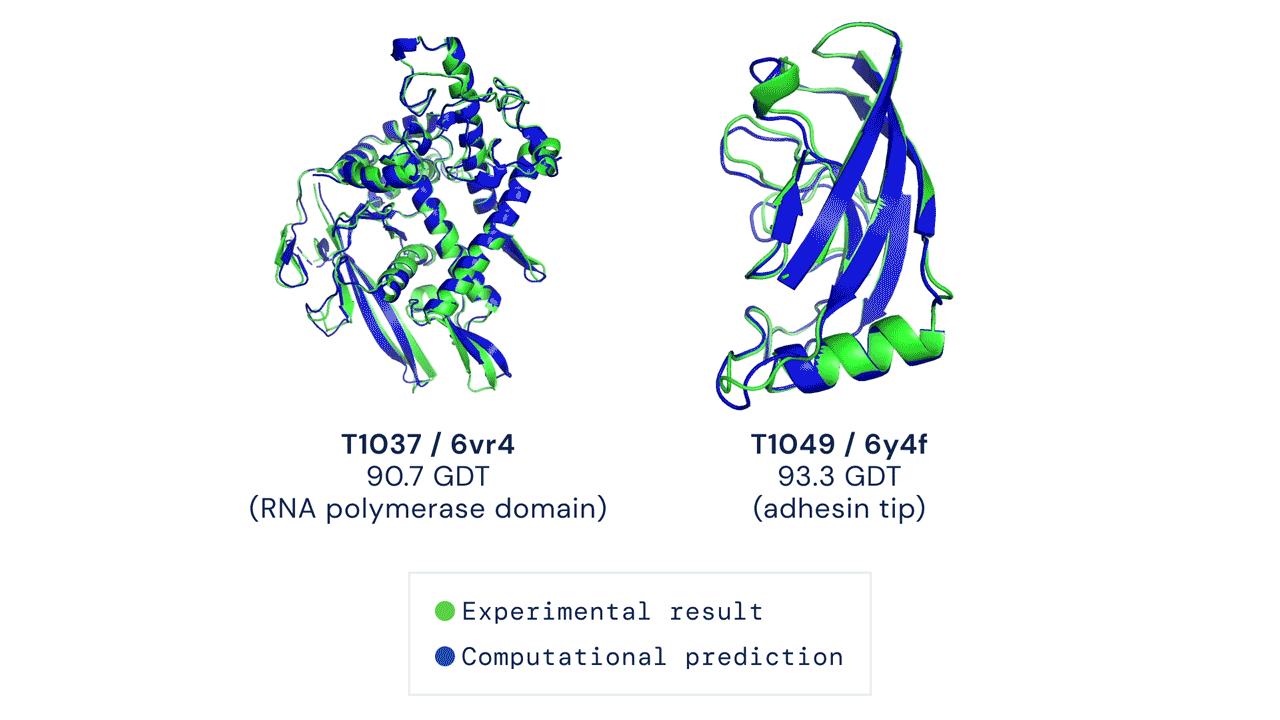 Två exempel på proteinstrukturer förutsagda av AlphaFold (i blått) jämfört med experimentella resultat (i grönt). Bild: DeepMind
Två exempel på proteinstrukturer förutsagda av AlphaFold (i blått) jämfört med experimentella resultat (i grönt). Bild: DeepMind Det finns för närvarande cirka 180 000 proteinstrukturer tillgängliga i det offentliga området, alla producerade med experimentella metoder och tillgängliga via Protein Data Bank. DeepMind släpper förutsägelser för strukturen av cirka 350 000 proteiner i 20 olika organismer, inklusive djur som möss och fruktflugor och bakterier som E. coli. (Det finns en viss överlappning mellan DeepMinds data och befintliga proteinstrukturer, men exakt hur mycket som är svårt att kvantifiera på grund av modellernas karaktär.) Mest betydelsefullt innehåller release förutsägelser för 98 procent av alla humana proteiner, cirka 20 000 olika strukturer, som kollektivt är kända som det humana proteomet. Det är inte den första offentliga datamängden av humana proteiner, men den är den mest omfattande och korrekta.
Om de vill kan forskare ladda ner hela det mänskliga proteomet för sig själva, säger AlphaFolds tekniska ledare John Jumper. “Det finns ett HumanProteome.zip effektivt, jag tror att det är ungefär 50 gigabyte i storlek”, säger Jumper till The Verge. “Du kan sätta den på en flash-enhet om du vill, men det skulle inte göra dig mycket bra utan en dator för analys!”
“vem som helst kan använda den för vad som helst”
Efter att ha lanserat den här första delen av data planerar DeepMind att fortsätta lägga till i butiken med proteiner, som kommer att underhållas av Europas flaggskeppsliv för biovetenskap, European Molecular Biology Laboratory (EMBL). I slutet av året hoppas DeepMind att släppa förutsägelser för 100 miljoner proteinstrukturer, en dataset som kommer att vara ”transformativ för vår förståelse av hur livet fungerar”, enligt Edith Heard, generaldirektör för EMBL.
Uppgifterna kommer att vara gratis för alltid för både vetenskapliga och kommersiella forskare, säger Hassabis. “Vem som helst kan använda den för vad som helst”, konstaterade DeepMind VD vid en press briefing. “De behöver bara kreditera de personer som är inblandade i citatet.”
Fördelarna med proteinvikning
Att förstå ett proteins struktur är användbart för forskare inom en rad olika områden. Informationen kan hjälpa till att utforma nya läkemedel, syntetisera nya enzymer som bryter ner avfallsmaterial och skapa grödor som är resistenta mot virus eller extrema väder. Redan används DeepMinds proteinförutsägelser för medicinsk forskning, inklusive att studera hur SARS-CoV-2 fungerar, det virus som orsakar COVID-19.
”Det kommer definitivt att ha en enorm inverkan för det vetenskapliga samfundet”
Ny data kommer att påskynda dessa ansträngningar, men forskare noterar att det fortfarande tar mycket tid att göra denna information till verkliga resultat. “Jag tror inte att det kommer att bli något som förändrar sättet att behandla patienter inom året, men det kommer definitivt att få en enorm inverkan för det vetenskapliga samfundet,” Marcelo C. Sousa, professor vid University of Colorado's biochemistry department. , berättade The Verge.
Forskare måste vänja sig vid att ha sådan information till hands, säger DeepMind seniorforskare Kathryn Tunyasuvunakool. “Som biolog kan jag bekräfta att vi inte har någon spelbok för att titta på till och med 20 000 strukturer, så denna [mängd data] är oerhört oväntad,” sa Tunyasuvunakool till The Verge. “Att analysera hundratusentals strukturer – det är galet.”
Anmärkningsvärt dock producerar DeepMinds programvara förutsägelser av proteinstrukturer snarare än experimentellt bestämda modeller, vilket innebär att det i vissa fall kommer att behövas ytterligare arbete för att verifiera strukturen. DeepMind säger att det tillbringade mycket tid på att bygga noggrannhetsmått i sin AlphaFold-programvara, vilket rankar hur säker det är för varje förutsägelse.
:no_upscale()/cdn .vox-cdn.com/uppladdningar/chorus_asset/file/22732723/Proteins.jpg ) Exempel på proteinstrukturer förutsagda av AlphaFold. Bild: DeepMind
Exempel på proteinstrukturer förutsagda av AlphaFold. Bild: DeepMind Förutsägelser av proteinstrukturer är fortfarande mycket användbara. Att bestämma ett proteins struktur genom experimentella metoder är dyrt, tidskrävande och förlitar sig på mycket försök och fel. Det betyder att även en lågförtroende förutsägelse kan spara forskare år av arbete genom att peka dem i rätt riktning för forskning.
Helen Walden, professor i strukturbiologi vid University of Glasgow, berättar för The Verge att DeepMinds data kommer att “avsevärt underlätta” forskningsflaskhalsar, men att “det mödosamma, resursdrivande arbetet med att göra biokemi och biologisk utvärdering av till exempel läkemedelsfunktioner ”kvarstår.
Sousa, som tidigare har använt data från AlphaFold i sitt arbete, säger att för forskare kommer effekten att kännas omedelbart. “I vårt samarbete som vi hade med DeepMind hade vi en dataset med ett proteinprov som vi hade haft i tio år, och vi hade aldrig kommit så långt att utveckla en modell som passar”, säger han. “DeepMind gick med på att ge oss en struktur, och de kunde lösa problemet på 15 minuter efter att vi hade satt på det i tio år.”
Varför proteinvikning är så svårt
Proteiner är konstruerade av kedjor av aminosyror, som finns i 20 olika sorter i människokroppen. Eftersom vilket enskilt protein som helst kan bestå av hundratals enskilda aminosyror, som var och en kan vikas och vridas i olika riktningar, betyder det att en molekyls slutliga struktur har ett oerhört stort antal möjliga konfigurationer. En uppskattning är att det typiska proteinet kan vikas på 10 ^ 300 sätt – det är en 1 följt av 300 nollor.
Proteinfällning har varit en “stor utmaning” för biologi i årtionden
Eftersom proteiner är för små för att undersökas med mikroskop har forskare varit tvungna att indirekt bestämma deras struktur med dyra och komplicerade metoder som kärnmagnetisk resonans och röntgenkristallografi. Idén att bestämma strukturen för ett protein genom att helt enkelt läsa en lista över dess ingående aminosyror har länge teoretiserats men svårt att uppnå, vilket har lett till att många beskriver det som en ”stor utmaning” för biologin.
Under senare år har dock beräkningsmetoder – särskilt de som använder artificiell intelligens – föreslagit att sådan analys är möjlig. Med dessa tekniker utbildas AI-system på datamängder av kända proteinstrukturer och använder denna information för att skapa sina egna förutsägelser.
:no_upscale()/cdn.vox-cd /chorus_asset/file/22732721/Median_Free_Modelling_Accuracy.png ) DeepMinds AlphaFold-programvara har väsentligt ökat noggrannheten för beräkningsproteinfällning, vilket visas av dess prestanda i CASP-tävlingen. Bild: DeepMind
DeepMinds AlphaFold-programvara har väsentligt ökat noggrannheten för beräkningsproteinfällning, vilket visas av dess prestanda i CASP-tävlingen. Bild: DeepMind Många grupper har arbetat med detta problem i flera år, men DeepMinds djupa bänk av AI-talang och tillgång till datorresurser gjorde det möjligt att påskynda framstegen dramatiskt. Förra året tävlade företaget i en internationell proteinvikningstävling som kallades CASP och sprängde bort tävlingen. Dess resultat var så exakta att beräkningsbiologen John Moult, en av CASP: s grundare, sa att “i någon mening är problemet [med proteinvikning] löst.”
DeepMinds AlphaFold-program har uppgraderats sedan förra årets CASP-tävling och är nu 16 gånger snabbare. “Vi kan lägga ett genomsnittligt protein på några minuter, i de flesta fall sekunder”, säger Hassabis. Företaget släppte också den underliggande koden för AlphaFold förra veckan som öppen källkod, så att andra kan bygga på sitt arbete i framtiden.
Liam McGuffin, professor vid Reading University som utvecklade några av Storbritanniens ledande proteinfällningsprogram, berömde AlphaFolds tekniska glans, men noterade också att programmets framgång bygger på årtionden av tidigare forskning och offentliga data. “DeepMind har stora resurser för att hålla denna databas uppdaterad och de är bättre lämpade att göra detta än någon enskild akademisk grupp”, sa McGuffin till The Verge. “Jag tror att akademiker skulle ha kommit dit i slutändan, men det skulle ha varit långsammare eftersom vi inte har lika bra resurser.”
Varför bryr sig DeepMind?
Många forskare The Verge pratade med noterade DeepMinds generositet när de släppte dessa data gratis. När allt kommer omkring ägs labbet av Google-parent Alphabet, som har hällt stora mängder resurser på kommersiella sjukvårdsprojekt. DeepMind förlorar själv mycket pengar varje år, och det har rapporterats om spänningar mellan företaget och dess moderföretag i frågor som forskningsautonomi och kommersiell lönsamhet.
Hassabis berättar dock för The Verge att företaget alltid planerat att göra denna information fritt tillgänglig, och att detta är en uppfyllande av DeepMinds grundläggande etos. Han betonar att DeepMinds arbete används på många ställen på Google – “nästan vad du än använder, det finns en del av vår teknik som är en del av det under huven” – men att företagets primära mål alltid har varit grundläggande forskning.
“Det finns många sätt att uppnå värde.”
”Avtalet när vi förvärvades är att vi främst är här för att främja läget för AGI och AI-teknik och sedan använda det för att påskynda vetenskapliga genombrott”, säger Hassabis. “[Alfabetet] har många divisioner som fokuserar på att tjäna pengar”, tillägger han och noterar att DeepMinds fokus på forskning “ger alla möjliga fördelar, när det gäller prestige och goodwill för det vetenskapliga samfundet. Det finns många sätt att uppnå värde. ”
Hassabis förutspår att AlphaFold är ett tecken på de kommande sakerna – ett projekt som visar den enorma potentialen med artificiell intelligens för att hantera röriga problem som mänsklig biologi.
“Jag tror att vi befinner oss i ett riktigt spännande ögonblick”, säger han. “Under det kommande decenniet hoppas vi och andra inom AI-fältet att producera fantastiska genombrott som verkligen kommer att påskynda lösningar på de riktigt stora problemen vi har här på jorden.”